【标题】Deep embedding and alignment of protein sequences
【作者团队】Felipe Llinares-L´opez, Quentin Berthet, Mathieu Blondel, Olivier Teboul, Jean-Philippe Vert
【发表时间】2021/07/01
【机 构】谷歌
【论文链接】https://doi.org/10.1101/2021.11.15.468653
【代码链接】https://github.com/google-research/google-research/tree/master/dedal
蛋白质序列比对是大多数研究蛋白质结构和功能的生物信息学方法的一个关键组成部分。然而,对高度多样性的序列进行比对仍然是一项艰巨的任务,目前的算法往往不能准确执行,导致许多蛋白质注释的不完善。本文利用深度学习在语言建模和可微分编程方面的最新进展,提出了DEDAL,一个用于比对蛋白质序列并检测同源物的灵活的模型。DEDAL是一个基于预训练的模型,它通过观察原始蛋白质序列和正确排列的大型数据集来学习排列序列。一旦经过训练,本文表明DEDAL比现有方法在远程同源物上的比对正确率提高了2-3倍,并能更好地将远程同源物与进化上不相关的序列区分开来,为改善结构和功能基因组学中许多依赖序列比对的下游任务提供了方案。
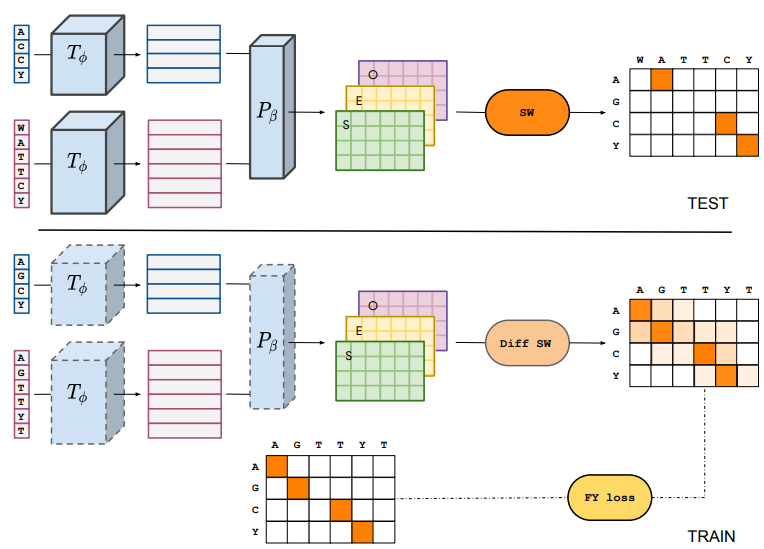
上图展示了DEDAL(Deep Embedding and Differentiable ALignment)的概述。它建立在标准的SW算法之上,是一个平滑的SW算法的变体。它可以有效地找到两个序列之间的最佳比对,但提供了一个灵活的SW算法使用的评分函数的参数化,以适应每个序列对和每个序列中的每个位置。DEDAL通过在具有已知比对的序列对和一大组原始蛋白质序列中运行SW局部对齐算法与特定的参数(gap开放O,gap扩展E,替换分数S)来对齐两个序列,这些参数取决于输入序列,通过每个序列的Transformer编码Tφ,然后是参数器Pβ,将连续表示转化为SW需要的参数矩阵。Tφ和Pβ都依赖于参数φ和β,这些参数在训练时从大型原始序列语料库中预训练学习语言模型Tφ,结合已知对齐方式的序列对,通过SW的连续可微变体和特定的对齐损失函数进行端到端梯度优化,共同学习Tφ和Pβ。
一旦经过训练,DEDAL就会产生专门为每一对新的序列计算的间隙和替换的评分矩阵。此外,间隙和替换的分数是有背景的:对于每一对位置,它们取决于要对齐的完整序列。然后用一个标准的SW算法使用这些参数计算出最佳的排列。本文表明,DEDAL可以在带有加速器的现代硬件上有效地训练。一旦训练完成,DEDAL与标准SW相比,为远程同源物预测的对准质量提高了2-3倍,并产生了一个能更准确检测远程同源物的对准分数。
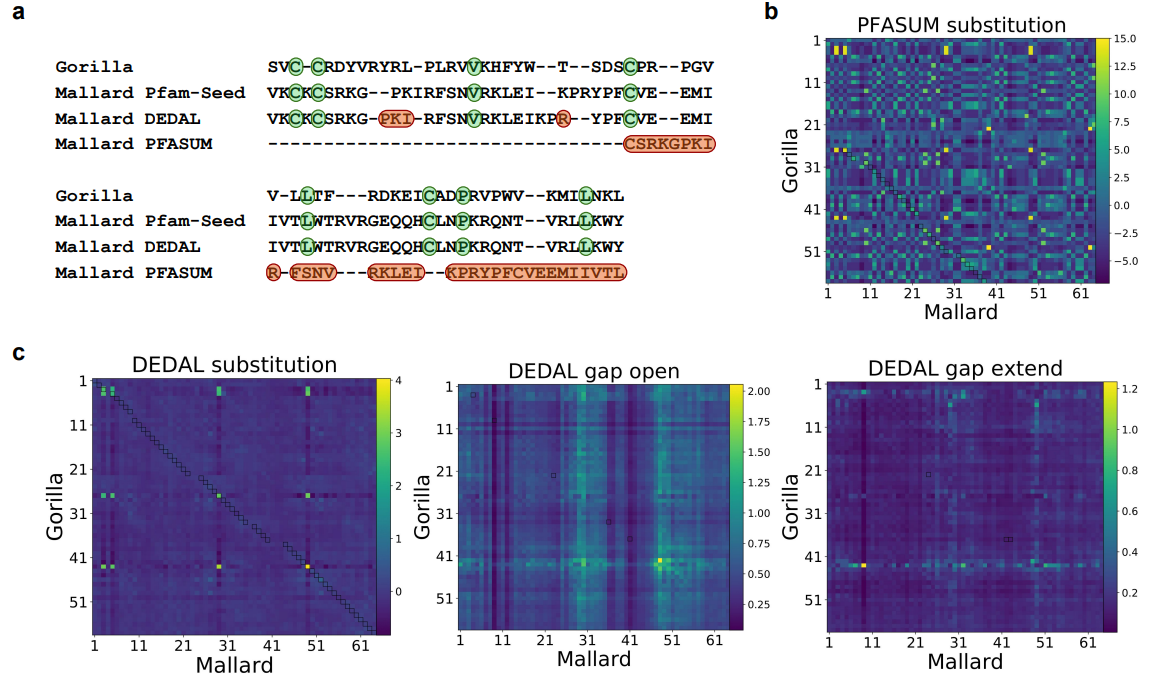
上图展示了来自Pfam-A种子的两个蛋白域序列比对例子。
a. 分别从Pfam-A种子数据库(第二行)、DEDAL预测(第三行)和用PFASUM70替代矩阵预测(第四行)进行的比对。本文显示了Pfam-A种子和DEDAL对准的两个序列中的所有残基,但没有显示PFASUM的序列中对准的上游和下游的未对准残基。绿色突出显示的残基对应于正确对齐的保守残基,而红色显示的残基对应于预测对齐和Pfam-A种子对齐之间的差异。
b. 来自PFASUM替代矩阵的所有残基对之间的替代分数。
c. 由DEDAL预测的SW参数。
在技术方面,本文探索了两种方法来创建一个可区分的SW对齐模块,需要在 "学习对齐 "任务中训练DEDAL的参数,使用平滑技术或扰动技术;本文发现两者在性能上没有明显区别,并在最终的DEDAL模型中实施了基于扰动的方法。关于用于训练DEDAL的排列组合,本文发现,当本文希望DEDAL能够预测准确的局部排列时,使用Pfam扩展域而不是Pfam域是有益的。在遮蔽语言建模任务中预训练DEDAL时,将与分布外家族相关的序列从 "蛋白质宇宙 "中排除,导致远程同源物的性能略有下降,尽管相对于与基线的性能差距来说并不明显。
关于端到端联合训练变换器和参数器的策略,本文发现这确实明显优于更经典的两步策略,即首先在屏蔽的语言建模任务中训练变换器编码器,然后通过保持变换器固定在 "学习对齐 "任务中训练参数器。这表明,一个通用的语言模型,如ESM,是不够的,至少应该进行微调,以达到对齐的最佳性能。
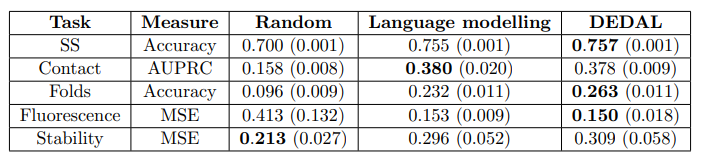
上图展示了学习的嵌入在下游任务的应用情况。本文通过简单地训练一个模型来评估与上下文相关的嵌入的好处,在这个模型中,替换成本被限制为只取决于要对齐的氨基酸;不难看出,本文观察到这个模型的性能有很大的下降,达到了与 "对准 "中表现最好的替换矩阵差不多的性能。





评论
沙发等你来抢